들어가기전...
SVM은 실무보다 이론을 위한 모델의 느낌입니다. 커널트릭, 라그랑주 승수, 쌍대 문제등 수학적으로 어려운 개념이 많이 나오고 이해가 매우 어렵지만, 공부해야할 것은 매우 많고 최근 캐글등에서 거의 사용되지 않는 것 같습니다.
XGB나 LightGBM, 딥러닝 등에 비해, 학습 시간은 매우 오래 걸리고 성능도 그다지 좋지 않은 느낌입니다.
90년대에서 2000년 초반까지 성능이 매우 좋았던 모델인데, 학술용의 느낌만 남아있습니다.
시험에 나오는 용어의 개념이나, 하이퍼 파라미터 정도만 이해하는 편이 좋을것 같아, 교재에 나왔던 용어정도만 정리하고자 합니다. 수식 유도등은 너무 시간 낭비로 보여집니다.
5.1 선형 SVM(Support Vector Machine)
SVM은 클래스들을 구분하는 결정 경계(Decision Boundary)로부터, 가장 가까운 다른 클래스의 데이터(서포트 벡터)를 지나는 두 마진 경계(Margin Boundaries, 또는 Support Vector Hyperplanes) 사이의 거리(마진)를 최대화하는 초평면을 찾는 모델입니다.
결정 경계(Decision Boundary)
두 클래스를 구분할 때, 하나의 클래스는 $w^T x + b > 0$, 다른 클래스는 $w^T x + b < 0$ 이 되는 영역으로 나뉜다고 할 때, 두 클래스를 나누는 경계인 $w^T x + b = 0$ 을 결정 경계(Decision Boundary) 라고 합니다.
초평면은 고차원 공간에서 w벡터와 직교하는 공간입니다.
마진 경계 (Margin Boundaries)
$w^T x + b = +1, \quad w^T x + b = -1$
마진을 정규화된 거리 1로 생각하면, margin mm을 가지는 두 개의 초평면을 다음과 같이 설정합니다:
마진 (Margin)
결정 경계( $w^T x + b = 0$ )에서 마진 경계( $w^T x + b = \pm 1$ )까지의 거리는 다음과 같습니다:두 마진 경계 사이 거리 : $\text{margin} = \frac{2}{\|w\|}$
여기서 $\|w\|$은 벡터 의 L2 노름입니다.
SVM은 마진을 최대화 하는 것이기 때문에, 분모의 w를 최소화 해야합니다.
계산의 편의성을 위해 마진을 최대화 하기 위한 목적 함수는 미분을 고려하여 아래를 최소화하는것을 목표로 합니다.
$\frac{1}{2} || w||^2$
미분을 위해 형태 변경되었습니다.
두 벡터를 완변하게 나누는 것을 하드 마진 분류라고 하는데, 아래와 같은 수식으로 표현됩니다.
$min \frac{1}{2} w^Tw$
하드마진과 소프트 마진
(1) 하드마진(Hard Margin)
클래스들을 오차없이 완벽히 마진을 최대화하여 분리하는 경우
제한조건 : $y^{(i)}(w^Tx^{(i)}+b)≥1$
목적함수 : $min_{ w,b}\frac{1}{2}||w||^2$
현실 세계의 데이터는 완벽하게 분리되지 않으며, 이상치등이 있으면 매우 불안정해집니다.
(2) 소프트마진(Soft Margin)
클래스들을 약간의 오차를 허용하여 분리하는 경우입니다.제약조건
$subject\ to\ y^{(i)} (w^T x^{(i)} + b) >= 1 - ξ_i, \ for\ all\ i
\ ξ_i >= 0,\ for\ all\ i
$
목적함수
$minimize (1/2) * ||w||^2 + C * sum_{i=1}^{n} ξ_i$
여기서, C는 오차 허용 패널치를 조절하는 하이퍼 파라미터입니다.
C가 크면 하드마진 처럼 오차를 허용하지 않고, C가 작으면 오차를 허용하고 마진을 넗게 만듭니다.
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.svm import LinearSVC
from sklearn.preprocessing import StandardScaler
from sklearn.pipeline import make_pipeline
# ===== 1. 데이터 준비 =====
iris = datasets.load_iris()
X = iris["data"][:, (2, 3)] # petal length, petal width
y = (iris["target"] == 0).astype(int) # setosa(0) vs others(1)
# versicolor(1) vs setosa(0)만 선택
X = X[(iris['target'] == 0) | (iris['target'] == 1)]
y = (iris['target'][(iris['target'] == 0) | (iris['target'] == 1)])
# ===== 2. 하드/소프트 마진 SVM 학습 =====
svm_clf_hard = make_pipeline(StandardScaler(), LinearSVC(C=1e10, loss="hinge", max_iter=10000, dual=True))
svm_clf_hard.fit(X, y)
svm_clf_soft = make_pipeline(StandardScaler(), LinearSVC(C=0.1, loss="hinge", max_iter=10000, dual=True))
svm_clf_soft.fit(X, y)
# ===== 3. 결정 경계와 마진 경계 그리기 함수 =====
def plot_svm_subplot(ax, svm_clf_pipeline, X, y, title):
svm_clf = svm_clf_pipeline.named_steps['linearsvc']
scaler = svm_clf_pipeline.named_steps['standardscaler']
X_scaled = scaler.transform(X)
ax.scatter(X_scaled[:, 0], X_scaled[:, 1], c=y, cmap="bwr", s=50, edgecolors="k")
xlim = ax.get_xlim()
ylim = ax.get_ylim()
xx = np.linspace(xlim[0], xlim[1], 30)
yy = np.linspace(ylim[0], ylim[1], 30)
YY, XX = np.meshgrid(yy, xx)
xy = np.vstack([XX.ravel(), YY.ravel()]).T
Z = svm_clf.decision_function(xy).reshape(XX.shape)
# 결정 경계와 마진 경계
ax.contour(XX, YY, Z, colors="k", levels=[-1, 0, 1], alpha=0.7,
linestyles=["--", "-", "--"])
# 서포트 벡터 근처 포인트 표시
decision_function = svm_clf.decision_function(X_scaled)
close = np.abs(decision_function) <= 1.2
ax.scatter(X_scaled[close, 0], X_scaled[close, 1], s=150, facecolors='none', edgecolors='k')
ax.set_xlabel("Petal length (scaled)")
ax.set_ylabel("Petal width (scaled)")
ax.set_title(title)
ax.set_xlim(xlim)
ax.set_ylim(ylim)
ax.grid(True)
# ===== 4. 하드 마진과 소프트 마진을 가로로 표시 =====
fig, axes = plt.subplots(1, 2, figsize=(16, 6))
plot_svm_subplot(axes[0], svm_clf_hard, X, y, "Hard Margin SVM")
plot_svm_subplot(axes[1], svm_clf_soft, X, y, "Soft Margin SVM")
plt.tight_layout()
plt.show()
각 샘플이 결정 경계에서 얼마나 떨어져있는지 계산해서 Decision score를 출력할 수 있습니다.
svm_clf_hard.decision_function(X)array([-1.92789234, -1.92789234, -2.00094662, -1.85483806, -1.92789234,
-1.31783531, -1.73244525, -1.85483806, -1.92789234, -2.05028515,
-1.85483806, -1.78178378, -2.12333943, -2.34250228, -2.0740009 ,
-1.46394387, -1.61005243, -1.73244525, -1.5132824 , -1.65939096,
-1.70872949, -1.46394387, -2.22010946, -1.12238822, -1.56262093,
-1.78178378, -1.39088959, -1.85483806, -1.92789234, -1.78178378,
-1.78178378, -1.46394387, -2.05028515, -1.92789234, -1.85483806,
-2.0740009 , -2.00094662, -2.12333943, -2.00094662, -1.85483806,
-1.80549953, -1.80549953, -2.00094662, -0.9999954 , -1.17172675,
-1.73244525, -1.78178378, -1.92789234, -1.85483806, -1.92789234,
2.82826406, 2.87760259, 3.16981972, 2.121437 , 2.95065687,
2.48670841, 3.21915825, 1.02371575, 2.55976269, 2.24382981,
1.16982431, 2.65843975, 1.53509572, 2.82826406, 1.82921987,
2.60910122, 2.87760259, 1.60815 , 2.87760259, 1.65748853,
3.68310672, 2.121437 , 3.16981972, 2.43736988, 2.34059984,
2.60910122, 2.90131834, 3.63376819, 2.87760259, 1.16982431,
1.58443425, 1.31593288, 1.85293563, 3.51137537, 2.87760259,
3.07304969, 3.02371116, 2.41365413, 2.19449128, 2.121437 ,
2.21820703, 2.75520978, 1.92598991, 1.02371575, 2.26754556,
2.07209847, 2.26754556, 2.34059984, 1. , 2.19449128])
5.2 비선형 SVM
선형 회귀에서 다항식을 만들 때는 Polynomial Feature로 변환한 후 Linear Regression을 수행하여 고차원 선형 회귀를 합니다.
이 경우 feature 수가 다항식 차수만큼 늘어나므로 계산량이 많아집니다.
SVM에서는 커널 트릭(Kernel Trick) 이라는 기법을 사용하여, 고차원 변환 없이도 고차원 선형 분류 효과를 얻습니다.
커널 트릭 개념
- SVM은 초평면(hyperplane)으로 데이터를 분리합니다.
- 현실 데이터는 대부분 비선형 분포를 가지므로, 선형 초평면으로 분리하기 어렵습니다.
- 데이터를 고차원으로 변환하면 분리가 쉬워지지만, 직접 변환하면 계산량이 폭발적으로 증가합니다.
- 커널 트릭은 고차원 변환 없이, 데이터 간 유사도(similarity) 를 계산하여 이 문제를 해결합니다.
- SVM에서는 유사도 함수의 일종으로 커널 함수를 사용하여, 고차원 공간의 내적을 대신 계산해서 포인트간 유사도(similarity)를 계산합니다.
커널 트릭은 다음처럼 작동합니다:
$K(x,x')=(ϕ(x), ϕ(x'))$
- : 입력 를 고차원으로 매핑하는 함수
- K(x, x') : 고차원 공간에서의 내적을 직접 계산하지 않고, 커널 함수로 대신 계산
SVM 대표 커널 함수
| 커널 종류 | 수식 | 특징 |
| 선형 커널 | $K(x, x') = x^T x'$ | 기본 SVM (선형 결정 경계) |
| 다항 커널 | $K(x, x') = ( \gamma x^T x' + r)^d$ | 곡선 결정 경계 생성 |
| RBF 커널 (가우시안) | $K(x, x') = \exp(-\gamma \|x - x'\|^2)$ | 매우 복잡한 비선형 결정 경계 생성 |
| 시그모이드 커널 (Sigmoid) | $K(x,x′)=tanh(γx^Tx′+r)$ | 시그모이드 결정 경계 생성 |
가우시안 커널(RBF) 설명
RBF 커널 수식:
$K(x,x′)=exp(−γ||x−x′||^2)$
- 두 점 x와 x′가 가까우면 K(x, x')가 1에 가까워지고,
- 멀면 K(x, x')는 0에 가까워집니다.
- 비슷한 데이터는 높은 유사도를 가지며, 다르면 유사도가 낮아져 비선형 결정 경계가 자연스럽게 형성됩니다.
커널 계산 예
두 개의 2차원 벡터 , x′=(2,3)
(1) 선형 커널 계산:
$K(x, x') = x^T x' = (1)(2) + (2)(3) = 2 + 6 = 8$
(2) 다항 커널 계산 : , d=2 (2차 다항 커널) 로 설정하면:
$K(x,x′)=( (1)(2)+(2)(3) +1)^2=9^2=81$
(3) RBF 커널 계산 (γ = 0.5라고 가정):
$K(x, x') = \exp\left( -0.5 \times ((1-2)^2 + (2-3)^2) \right) = \exp\left( -0.5 \times (1 + 1) \right) = \exp(-1) ≈ 0.3679$
비선형 SVM 하이퍼 파라미터
| 파라미터 | 의미 | 영향 |
| γ (gamma) |
유사도 반경 조절 γ가 크면 유사도가 0에 가까워져 결정경계가 복잡해지고, 작으면 멀리 떨어진 데이터도 유사한걸로 판단, 결정 경계가 부드럽고 단순해집니다. |
복잡성 제어 (RBF, Poly, Sigmoid) |
| degree | 다항 커널에서 다항식 차수 결정 | 복잡성 제어 (Poly 전용) |
| coef0 | 내적에 상수항 추가( 수식의 r에 해당) | 비선형성 조정 (Poly, Sigmoid) |
| C | 오차 패널티 조절 | 과적합, 과소적합 조절 |
import numpy as np
import matplotlib.pyplot as plt
from sklearn.svm import SVC
from sklearn.preprocessing import StandardScaler
from sklearn.pipeline import make_pipeline
from sklearn.datasets import make_moons
# ===== 1. 데이터 준비 =====
X, y = make_moons(n_samples=100, noise=0.15, random_state=42)
# ===== 2. 보조 함수 정의 =====
def plot_dataset(X, y, axes):
plt.plot(X[y == 0, 0], X[y == 0, 1], "bs", label="Class 0")
plt.plot(X[y == 1, 0], X[y == 1, 1], "g^", label="Class 1")
plt.axis(axes)
plt.grid(True)
plt.xlabel("$x_1$", fontsize=14)
plt.ylabel("$x_2$", fontsize=14, rotation=0)
plt.legend()
def plot_predictions(clf, axes):
x0s = np.linspace(axes[0], axes[1], 100)
x1s = np.linspace(axes[2], axes[3], 100)
x0, x1 = np.meshgrid(x0s, x1s)
X_new = np.c_[x0.ravel(), x1.ravel()]
y_pred = clf.predict(X_new).reshape(x0.shape)
plt.contourf(x0, x1, y_pred, cmap=plt.cm.brg, alpha=0.2)
# ===== 3. SVM 모델 학습 =====
gamma1, gamma2 = 0.1, 5
C1, C2 = 0.001, 1000
hyperparams = (gamma1, C1), (gamma1, C2), (gamma2, C1), (gamma2, C2)
svm_clfs = []
for gamma, C in hyperparams:
svm_clf = make_pipeline(
StandardScaler(),
SVC(kernel="rbf", gamma=gamma, C=C)
)
svm_clf.fit(X, y)
svm_clfs.append(svm_clf)
# ===== 4. 시각화 =====
fig, axes = plt.subplots(2, 2, figsize=(10, 7), sharex=True, sharey=True)
for idx, svm_clf in enumerate(svm_clfs):
ax = axes[idx // 2, idx % 2]
plt.sca(ax)
plot_predictions(svm_clf, [-1.5, 2.45, -1, 1.5])
plot_dataset(X, y, [-1.5, 2.45, -1, 1.5])
gamma, C = hyperparams[idx]
ax.set_title(f"gamma={gamma}, C={C}")
plt.tight_layout()
plt.show()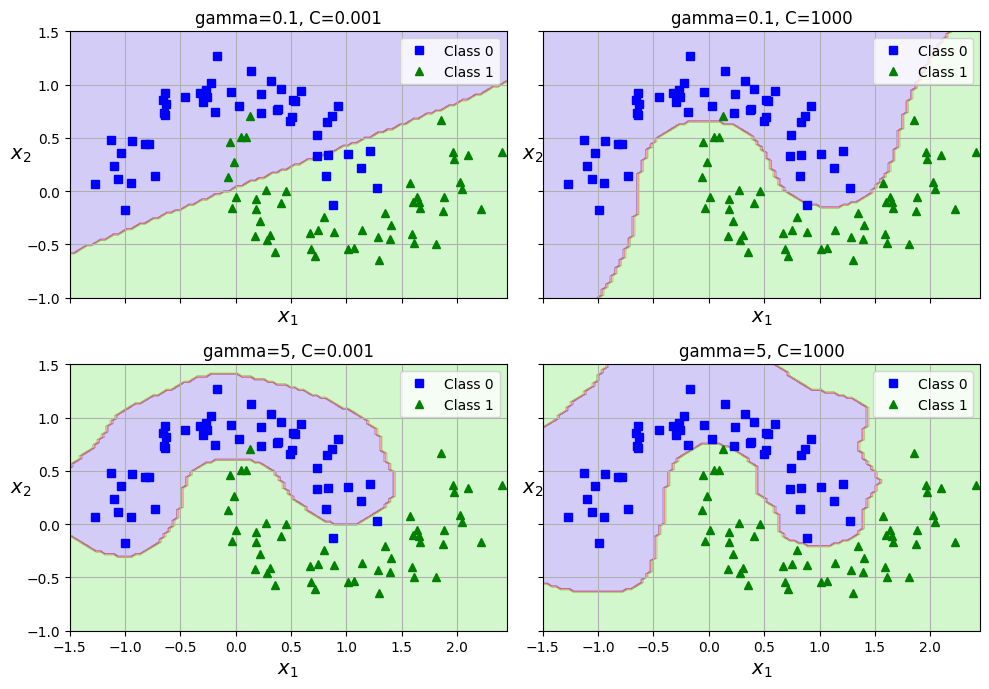
5.3 SVM 회귀
SVM 회귀(SVR)는 분류 문제처럼 마진 경계를 최대화하는 대신, 마진 경계(폭 ε) 안에 최대한 많은 데이터 포인트를 포함시키는 것을 목표로 합니다.
이 마진 폭을 하이퍼파라미터 ε(epsilon) 으로 조정합니다.
$| w^Tx + b - y | ≤ ϵ$
분류에서는 마진을 최대화 했지만, 회귀에서는 다음을 최소화 합니다.
$min = \frac{1}{2}||w||^2$
규제항을 포함한 목적함수는
$min_{w,b,ξ,ξ*} = \frac{1}{2}||w||^2 + C \sum_{i=1}^{n} (ξ_i+ξ*_i)$
- 예측값: $w^T x + b$
- 실제값: y
- 목 표: |예측 - 실제| <= ε
- 초 과: 초과한 만큼 ξ, ξ*로 벌점
import numpy as np
import matplotlib.pyplot as plt
from sklearn.svm import LinearSVR, SVR
from sklearn.preprocessing import StandardScaler
from sklearn.pipeline import make_pipeline
# ===== 1. 데이터 준비 =====
# Linear SVR용 데이터
np.random.seed(42)
X_lin = 2 * np.random.rand(50, 1)
y_lin = 4 + 3 * X_lin[:, 0] + np.random.randn(50)
# Polynomial SVR용 데이터
X_poly = 2 * np.random.rand(50, 1) - 1
y_poly = 0.2 + 0.1 * X_poly[:, 0] + 0.5 * X_poly[:, 0]**2 + np.random.randn(50) / 10
# ===== 2. 보조 함수 =====
def find_support_vectors(svm_reg, X, y):
y_pred = svm_reg.predict(X)
epsilon = svm_reg[-1].epsilon
off_margin = np.abs(y - y_pred) >= epsilon
return np.argwhere(off_margin)
def plot_svm_regression(svm_reg, X, y, axes):
x1s = np.linspace(axes[0], axes[1], 100).reshape(-1, 1)
y_pred = svm_reg.predict(x1s)
epsilon = svm_reg[-1].epsilon
plt.plot(x1s, y_pred, "k-", linewidth=2, label=r"$\hat{y}$", zorder=-2)
plt.plot(x1s, y_pred + epsilon, "k--", zorder=-2)
plt.plot(x1s, y_pred - epsilon, "k--", zorder=-2)
if hasattr(svm_reg, "_support"):
plt.scatter(X[svm_reg._support, 0], y[svm_reg._support], s=180,
facecolors='#AAA', edgecolors='k', zorder=-1)
plt.plot(X, y, "bo")
plt.xlabel("$x_1$")
plt.axis(axes)
plt.grid(True)
# ===== 3. 모델 학습 =====
# LinearSVR 모델 2개
svm_reg1 = make_pipeline(StandardScaler(), LinearSVR(epsilon=0.5, dual=True, random_state=42))
svm_reg2 = make_pipeline(StandardScaler(), LinearSVR(epsilon=1.2, dual=True, random_state=42))
svm_reg1.fit(X_lin, y_lin)
svm_reg2.fit(X_lin, y_lin)
svm_reg1._support = find_support_vectors(svm_reg1, X_lin, y_lin)
svm_reg2._support = find_support_vectors(svm_reg2, X_lin, y_lin)
# Polynomial SVR 모델 2개
svm_poly1 = make_pipeline(StandardScaler(), SVR(kernel="poly", degree=2, C=0.01, epsilon=0.05))
svm_poly2 = make_pipeline(StandardScaler(), SVR(kernel="poly", degree=2, C=100, epsilon=0.05))
svm_poly1.fit(X_poly, y_poly)
svm_poly2.fit(X_poly, y_poly)
svm_poly1._support = find_support_vectors(svm_poly1, X_poly, y_poly)
svm_poly2._support = find_support_vectors(svm_poly2, X_poly, y_poly)
# ===== 4. 시각화 (2×2로) =====
fig, axes = plt.subplots(2, 2, figsize=(12, 8), sharey=False)
# 좌상: LinearSVR (ε=0.5)
plt.sca(axes[0, 0])
plot_svm_regression(svm_reg1, X_lin, y_lin, [0, 2, 3, 11])
plt.title(f"LinearSVR, ε={svm_reg1[-1].epsilon}")
# 우상: LinearSVR (ε=1.2)
plt.sca(axes[0, 1])
plot_svm_regression(svm_reg2, X_lin, y_lin, [0, 2, 3, 11])
plt.title(f"LinearSVR, ε={svm_reg2[-1].epsilon}")
# 좌하: Polynomial SVR (C=0.01)
plt.sca(axes[1, 0])
plot_svm_regression(svm_poly1, X_poly, y_poly, [-1, 1, 0, 1])
plt.title(f"PolySVR, degree={svm_poly1[-1].degree}, C={svm_poly1[-1].C}")
# 우하: Polynomial SVR (C=100)
plt.sca(axes[1, 1])
plot_svm_regression(svm_poly2, X_poly, y_poly, [-1, 1, 0, 1])
plt.title(f"PolySVR, degree={svm_poly2[-1].degree}, C={svm_poly2[-1].C}")
plt.tight_layout()
plt.show()
5.4 SVM 이론
| 마진 최대화 | SVM의 기본 목표는 마진(Margin) 을 최대화하는 결정 경계(초평면)를 찾는 것 |
| Soft Margin | 완벽하게 분리할 수 없는 경우 오차를 일부 허용(Soft Margin Classification) 하고 패널티를 부여 |
| Slack 변수 (ξ) | 오차가 발생한 정도를 나타내는 변수. 결정 경계에 가까운 샘플이나 잘못 분류된 샘플을 표현 |
| 목적 함수 | 마진을 넓히는 것($\frac{1}{2}\|w\|^2$ 최소화) + Slack 변수에 패널티 부과 |
| 하이퍼파라미터 C | 오차(슬랙 변수)에 부과하는 패널티의 크기를 조정. C가 크면 오차 허용을 줄이고, 작으면 오차를 더 허용 |
| 커널 트릭 | 데이터 포인트를 고차원으로 변환하지 않고, 커널 함수를 이용해 고차원 내적을 대신 계산 |
| 대표 커널 함수 | 선형 커널, 다항 커널(Polynomial), RBF(가우시안) 커널, 시그모이드 커널 |
| SVR (Support Vector Regression) | 분류가 아니라 회귀 문제에 SVM을 적용. ε-튜브 안에 데이터가 들어오도록 예측선 생성 |

5.5 쌍대문제
원문제(Primal Problem)과 쌍대문제(Dual Problem)
제약이 있는 처음 정의된 최적화하려는 원문제(Primal Problem)가 있으면, 원문제를 변형해 다른 형태로 다시 표현한 쌍대문제(Dual Problem)으로 표형할 수 있습니다.
SVM에서는 비선형 계산 식 대신, 커널 트릭을 사용해 유사도를 구할 수 있습니다.
원문제
$min_{w,b} = \frac{1}{2}||w||^2$
원문제의 w,b를 직접 찾는 대신, 각 데이터 샘플마다 부여하는 가중치(α) 를 찾는 상대문제 형태로 부꿀수 있습니다.
쌍대문제
$max_a = \sum_{i} \alpha_i - \frac{1}{2} \sum_{i,j} \alpha_i \alpha_j y^i y^j (x^i)^T x^j$
SVM 흐름
(1) 기본 SVM: w 찾기
↓
(2) 쌍대 문제: alpha 찾기 (샘플 간 내적만 사용)
↓
(3) 커널 SVM: 내적 대신 커널 함수 K(x, x') 사용
↓
(4) 비선형 경계
참고 : SVM의 Loss함수
| 모델 | 기본loss | 설명 |
| SVC | hinge (Soft Margin) | 분류 |
| LinearSVC | squared hinge | 선형 분류 |
| SVR | ε-insensitive loss | 회귀 |
| LinearSVR | ε-insensitive loss | 선형 회귀 |
(1) Hinge loss : 정답방향으로 1이상 떨어져야 만족하는 Loss함수입니다. yf(x)가 1보다 크면 loss는 0이고, 1보다 작으면 선형으로 패널티가 증가합니다.

| 상태 | Loss값 | 설명 |
| $y \cdot f(x) \geq 1$ | 0 | 올바르게 분류 + 충분한 마진 확보 |
| $0 < y \cdot f(x) < 1$ | $1 - y \cdot f(x)$ | 올바르게 분류했지만 마진 부족 (패널티 부여) |
| $y \cdot f(x) \leq 0$ | $1 - y \cdot f(x)$ | 잘못 분류 (큰 패널티) |
(2) Squared Hinge Loss
Hinge Loss를 제곱한 버전입니다. 마진을 넘지 못한 정도를 제곱으로 패널티를 부과합니다.

(3) ε-insensitive loss : 오차가 ε 이내면 Loss=0, 넘으면 초과한 만큼 패털티 부과합니다.

'교제정리 > 핸즈온머신러닝' 카테고리의 다른 글
| 핸즈온 머신러닝 제7장 앙상블 학습과 랜덤 포레스트 (0) | 2025.05.01 |
|---|---|
| 핸즈온 머신러닝 제6장 결정트리 (0) | 2025.04.29 |
| 핸즈온 머신러닝 4장 모델 훈련 (0) | 2025.04.27 |
| 핸즈온 머신러닝 3장 분류 (0) | 2025.04.26 |
| 핸즈온 머신러닝 2장 – 머신러닝 프로젝트 처음부터 끝까지 (0) | 2025.04.20 |
